Klassifikation von MRT in Zusammenarbeit mit dem Kantonsspital Winterthur
Klassifikation von MRT in Zusammenarbeit mit dem Kantonsspital Winterthur
Projekt von: Cornelia Schmitz, Norbert Bräker
 |
 |
| Cornelia Schmitz | Norbert Bräker |
Einführung
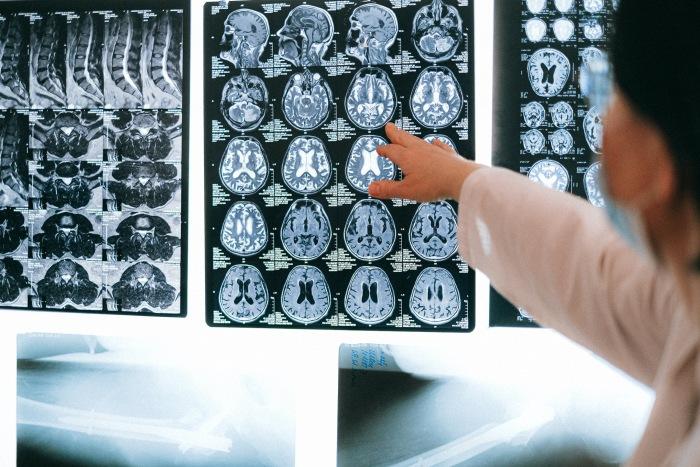
Das Ziel des Projekts war es, die Erkennung und Klassifizierung von Hirntumoren mit der Magnetresonanztomographie (MRT) mit Hilfe von Fortschritten in der Künstlichen Intelligenz, insbesondere neuronalen Netzen, zu automatisieren. Der Begriff "Hirntumor" umfasst sowohl die verschiedenen Arten, die sich direkt im Gehirn entwickeln, als auch Hirnmetastasen, d. h. Metastasen eines Tumors, der ursprünglich ausserhalb des Gehirns gewachsen ist. Die Erkennung von Hirntumoren und die Beurteilung der Art erfolgt heute durch Radiologen, die dies in der Regel mit Hilfe der Magnetresonanztomographie (MRT) zuverlässig durchführen können. Die Automatisierung dieses Prozesses hat jedoch den Vorteil, dass er als Zweitmeinung genutzt werden kann, wodurch das Risiko, einen kleinen Tumor zu übersehen oder falsch zu klassifizieren, weiter verringert wird. Ebenso kann die automatische Erkennung den Radiologen unterstützen, indem sie zum Beispiel die Lage mehrerer Metastasen genau dokumentiert, wenn diese mehrfach vorhanden sind (Quelle: SNFS).
Projektdetails
Cornelia Schmitz und Norbert Bräker bauten ein Klassifizierungsnetzwerk auf, um Bilder anhand von zwei Eigenschaften zu gruppieren: der Perspektive und der MR-Sequenz, die für die Erstellung jedes Bildes verwendet wurde. Die Aufgabe, die unsere Studenten erhielten, basiert auf einer früheren Arbeit von Paul Windisch, Physiker am Kantonsspital Winterthur und ehemaliger Data Science Student an der SIT Academy. Seine Idee war eine Pipeline von Netzwerken, die für die Erkennung von Hirntumoren verwendet werden. Als Input bekamen Cornelia und Norbert MR-Bilder und als Output sollten sie die Reihenfolge und Perspektive des Bildes vorhersagen.
Am Anfang dieser Pipeline von Netzwerken bestand die Aufgabe darin, neue Merkmale zu klassifizieren, die dann an das nächste Netzwerk weitergegeben werden. Die beiden Hauptmerkmale, mit denen unsere Studenten arbeiteten, waren Perspektive (Rückseite, Vorderseite, Vogelperspektive) und Sequenz. Die MR-Sequenz bestimmt, welche Gewebe heller oder dunkler erscheinen (auf dem Bild unten zu sehen). Die bereitgestellten Bilder wurden zunächst gruppiert und dann an spezialisierte Netzwerke weitergeleitet, um die Qualität der Tumorklassifizierung und -segmentierung zu verbessern.

Der Ansatz, der verwendet wurde, nennt sich Transfer Learning. Die Idee war, ein neuronales Netz zu verwenden, das zuvor mit Millionen von Bildern trainiert worden war. Der Vorteil dieses Ansatzes ist, dass man viel weniger Bilder benötigt, als wenn man das Netz von Grund auf neu trainieren müsste. Es wurden insgesamt 409 Bilder verwendet, die zunächst manuell ausgewählt und beschriftet werden mussten. Diese wurden dann in das zuvor trainierte Modell eingepflegt, dem zusätzliche Layers hinzugefügt wurden, damit das Netz speziell über MR-Bilder lernen konnte. Als Ergebnis erhielten unsere Studenten die Perspektive und die Reihenfolge der Bilder.
Ergebnisse des Transferlernens:

Da das Netz nun vollständig mit den verfügbaren Bildern trainiert ist, kann Zeit gespart werden, da die Bilder nicht mehr manuell ausgewählt werden müssen.
Am Anfang dieser Pipeline von Netzwerken bestand die Aufgabe darin, neue Merkmale zu klassifizieren, die dann an das nächste Netzwerk weitergegeben werden. Die beiden Hauptmerkmale, mit denen unsere Studenten arbeiteten, waren Perspektive (Rückseite, Vorderseite, Vogelperspektive) und Sequenz. Die MR-Sequenz bestimmt, welche Gewebe heller oder dunkler erscheinen (auf dem Bild unten zu sehen). Die bereitgestellten Bilder wurden zunächst gruppiert und dann an spezialisierte Netzwerke weitergeleitet, um die Qualität der Tumorklassifizierung und -segmentierung zu verbessern.

Der Ansatz, der verwendet wurde, nennt sich Transfer Learning. Die Idee war, ein neuronales Netz zu verwenden, das zuvor mit Millionen von Bildern trainiert worden war. Der Vorteil dieses Ansatzes ist, dass man viel weniger Bilder benötigt, als wenn man das Netz von Grund auf neu trainieren müsste. Es wurden insgesamt 409 Bilder verwendet, die zunächst manuell ausgewählt und beschriftet werden mussten. Diese wurden dann in das zuvor trainierte Modell eingepflegt, dem zusätzliche Layers hinzugefügt wurden, damit das Netz speziell über MR-Bilder lernen konnte. Als Ergebnis erhielten unsere Studenten die Perspektive und die Reihenfolge der Bilder.
Ergebnisse des Transferlernens:

Da das Netz nun vollständig mit den verfügbaren Bildern trainiert ist, kann Zeit gespart werden, da die Bilder nicht mehr manuell ausgewählt werden müssen.
Resultat
Zusammenfassend wurde ein Ansatz gefunden, welcher die Bilder korrekt klassifizieren, segmentieren und gruppieren kann. Während dieses Projekts wurden einige neue interessante Architekturen gefunden, die unsere Studenten weiter erforschen werden, um die Leistung der Modelle möglicherweise noch weiter zu verbessern. Die Klassifizierungsgenauigkeit des Transfer-Learning-Modells lag zwischen ~94% - 98% für Perspektive und Sequenz. Das Projekt insgesamt hat gezeigt, dass die Leistung eines Algorithmus zur Klassifizierung von Hirntumoren zufriedenstellend ist, aber noch nicht für die klinische Routineanwendung ausreicht. In weiteren Forschungsarbeiten soll nun untersucht werden, ob mit komplexeren Neuronalen Netzen auf den verwendeten Datensätzen bessere Ergebnisse erzielt werden können. Die in diesem Projekt geleistete Arbeit wird Paul Windisch bei seinen eigenen Forschungen zur automatischen Erkennung von Hirntumoren helfen, ein Projekt, das er zusammen mit Pascal Weber während seines Studiums an der SIT Academy begonnen hat und das nun vom SNF (Schweizerischer Nationalfonds) und der Innosuisse (Schweizerische Agentur für Innovationsförderung) finanziert wird.
Mehr Informationen findest du unter Cureus.
Mehr Informationen findest du unter Cureus.
Student
Cornelia Schmitz sagt:
Die SIT Academy hat mir geholfen, meine Fähigkeiten weiter zu entwickeln und gab mir das Selbstvertrauen, mich für interessante Aufgaben im Bereich der Datenwissenschaften zu bewerben. Durch die Erfahrungen, welche ich im Bootcamp gesammelt habe, konnte ich einen Job beim Technologie Start-up „Eyeware“ an Land ziehen.
Hello world